专题:线性方程组求解
简介
这里完整介绍一些求 的方法,特点是A矩阵的规模比较大。
正常来说,求解线性方程组可以通过求逆矩阵,高斯消元,克拉默法则(大学线代第一节课就学这个没用的东西,不会真的有人用吧,不会吧不会吧)等等方式去求解。
但是A很大的时候这个方法就失效了,这个时候一般用迭代法,本文以介绍迭代法为主。
这里解的方程组一定要正定,负定的矩阵解起来注定是失稳的。如果是正半定,或者说奇异矩阵(存在为0的特征值),那么解将会在某个子空间里而不唯一。
其实从二次型的视角就很容易看出:
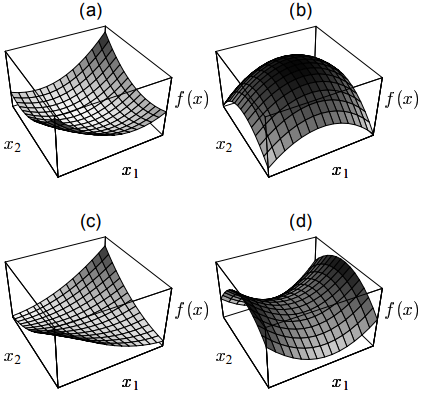
(请注意,本文中的推导仅仅是验证实验用,性能并非最优)
Jacobi 迭代与 Gauss-Seidel
Jacobi 迭代
推导过程:
- 我们把A矩阵分尸三块:
- 其中,D是对角线,L是上三角取负,U是下三角取负。
- 这样, 就可以写成
- 移动一下:
- 由于D是对角矩阵,可无成本求逆:
于是我们利用下列公式迭代更新:
即可求得解,至于为什么是这样,可看收敛性分析。
def jacobi_iteration(A,b,init_x,iters=10):
dim = init_x.shape[0]
x = init_x
result = np.zeros((iters+1,dim))
result[0,:] = x.reshape(-1)
diag_A = np.diag(A)
D_inv = np.diag(1.0/diag_A)
LU = np.diag(diag_A) - A
B = D_inv @ LU
D_inv_b = D_inv @ b
for i in range(iters):
x = B @ x + D_inv_b
result[i+1,:] = x.reshape(-1)
return result
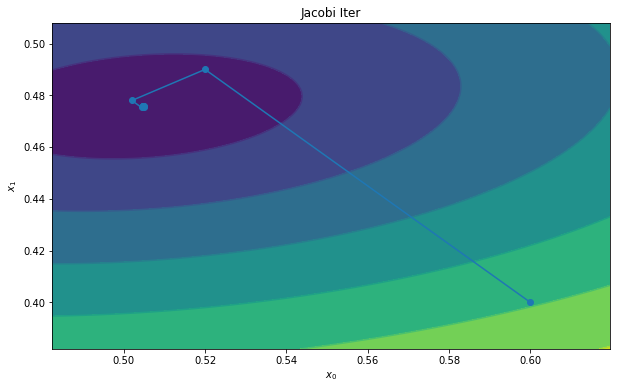
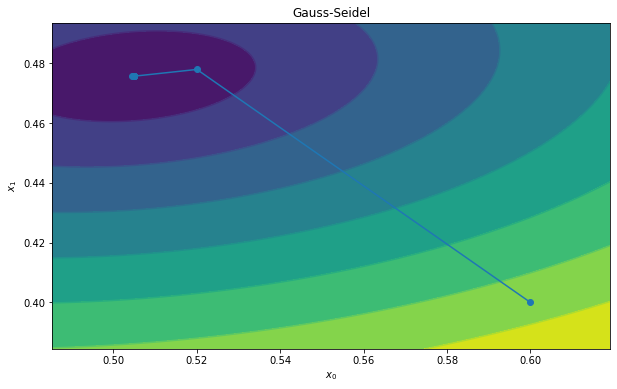
Gauss-Seidel
Jacobi迭代中,, 这里的 是向量,Jacobi迭代更新的时候是并行更新的。 但是Gauss-Seidel则是一个个元素更新的。
更新的时候会用到,同理,更新的时候会用到和。
(然而并没什么卵用,不必Jacobi快多少,还不能并行计算了。。。)
def gauss_seidel_iteration(A,b,init_x,iters=10):
dim = init_x.shape[0]
x = init_x
result = np.zeros((iters+1,dim))
result[0,:] = x.reshape(-1)
diag_A = np.diag(A)
D_inv = np.diag(1.0/diag_A)
LU = np.diag(diag_A) - A
B = D_inv @ LU
D_inv_b = D_inv @ b
for i in range(iters):
for d in range(dim):
x[d] = B[d,:] @ x + D_inv_b[d]
result[i+1,:] = x.reshape(-1)
return result
收敛性分析
如果矩阵A每一行的“除了对角线元素之外,其它元素的和”都要小于对角线元素的话,则这个矩阵一定可以被Jacobi迭代求解(我也不知为何如此,不过应该可以从这个条件推出谱半径小于1)。
还有一个就是B矩阵 () 的谱半径,谱半径等于最大的特征值。每个向量都可以被分解为特征向量的和(也就是表达在某个以特征向量为基的空间),如果谱半径大于1,那么求解的时候就会发散。谱半径不仅要小于1,而且要越小越好,这样收敛才够迅速。
而为什么我们关心B的谱半径呢?
我们站在神的视角,已经知道了真实的解x,也就是说x绝对满足: ,那么不妨设置误差
每一次迭代的时候,我们看作:
所以有:
我们知道 所以消去等号两侧的两项,得到:
所以,很直观的看到,只有谱半径小于1,才能保证误差不断减少,即所谓收敛。
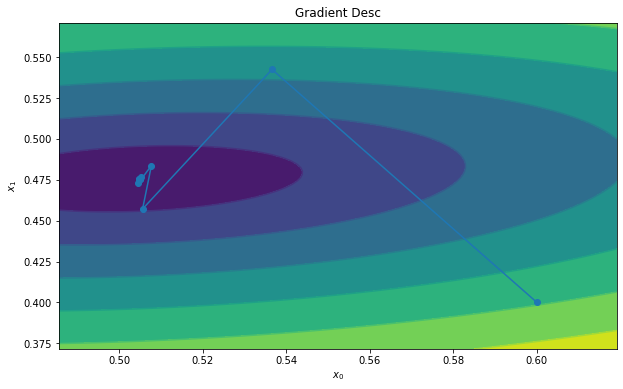
梯度下降
梯度下降迭代法的思路是优化
所以Loss Function就是
梯度方向有了,下面就是学习率了,和机器学习任务不一样,这里的学习率是可以有最优解的, 原理大概是如果沿着梯度方向前后走,并且记录路径上L的大小,那么我们会得到一个抛物线,抛物线是有极小值的啊!!!!
其中
或者说,走到新的点以后,其梯度应该和当前的梯度正交,这两种表述是等价的:
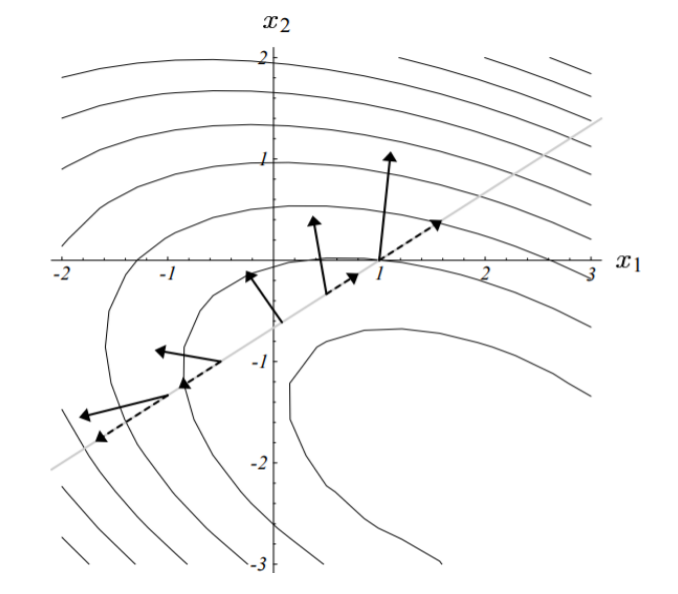
最优解是:
过程太难打暂略。
根据这个性质,梯度下降的算法将会以互相垂直的折线路径快速逼近最优解。
def gradient_desc(A,b,init_x,iters=10):
dim = init_x.shape[0]
x = init_x
result = np.zeros((iters+1,dim))
result[0,:] = x.reshape(-1)
for i in range(iters):
ax = A @ x
grad = ax - b
flat_x = x.reshape(-1)
lr = np.dot(flat_x,flat_x)/np.dot(ax.reshape(-1),flat_x)
x -= grad * lr
result[i+1,:] = x.reshape(-1)
return result
共轭梯度法
这个方法的思路是,不走回头路,如果我每一次新的搜索空间都和之前的所有搜索空间正交,那么可以更加高效。
但是求出一个正交的子空间(比如用施密特正交)复杂度他娘的是 啊。
Warm Start
在解微分方程(组)的时候,由于上一个时间片的状态和这个时间片的状态相差并不多,所以可以用上一个时间片的状态作为这个时间片的初始值输入,这样可以大大增加求解的效率。
MultiGrid
References
测试代码
import numpy as np
from matplotlib import pyplot as plt
def solver_visualizer(A:np.ndarray,b:np.ndarray,x:np.ndarray,steps:int=100):
"""
A: 2x2 matrix
b: 2x1 vector
x: nx2 vectors
"""
ax_min = x[:,0].min()
ax_max = x[:,0].max()
rx = ax_max - ax_min
ay_min = x[:,1].min()
ay_max = x[:,1].max()
ry = ay_max - ay_min
X,Y = np.meshgrid(
np.linspace(ax_min - 0.2 * rx,ax_max + 0.2 * rx,steps),
np.linspace(ay_min - 0.2 * ry,ay_max + 0.2 * ry,steps),
)
E = np.linalg.norm(
A @ np.array([X.reshape(-1),Y.reshape(-1)]) - b,
axis=0
).reshape(
X.shape
)
plt.figure(figsize=(10,6))
plt.contourf(X,Y,E)
plt.contour(X,Y,E)
plt.plot(x[:,0],x[:,1],'-o')
plt.show()
A = np.array([[1,0.2],[-0.3,2]])
b = np.array([0.6,0.8]).reshape(2,1)
init_x = b / np.diag(A).reshape(2,1)
# Example
solver_visualizer(A,b,gauss_seidel_iteration(A,b,init_x,10))